이번 포스팅에서는 클러스터링(군집화)이 잘되었는지 정량적으로 확인할 수 있는 평가 지표로 Dunn Index를 소개하려고 한다. 또한 Dunn Index를 파이썬(Python)으로 구현해보고자 한다.
이 곳은 꽁냥이가 머신러닝을 공부한 내용을 정리하는 곳입니다.
이 포스팅에서는 수식을 포함하고 있습니다. 티스토리 피드에서는 수식이 제대로 표시되지 않을 수 있으니 웹브라우저 또는 모바일 웹브라우저로 보시길 바랍니다.
1. 클러스터링(군집화) 평가의 필요성
왜 클러스터링(군집화) 결과를 평가해야 할까?
1) 클러스터링(군집화) 알고리즘의 성능을 비교하기 위해서이다.
여러 다른 클러스터링 알고리즘을 이용하여 클러스터링 결과를 얻었다고 하자. 결과가 모두 같다면 문제가 되지 않는다. 만약 다른 경우에는 어떤 클러스터링 결과를 선택해야 하는지에 대한 문제가 생길 수 있는데 이때 각 알고리즘 별로 점수(지표)를 매겨 성능을 비교하고 높은 성능을 갖는 클러스터링 알고리즘을 선택할 수 있다.
2) 클러스터 개수를 선정하기 위한 기준

하나의 클러스터링(군집화) 알고리즘에는 보통 클러스터 개수를 사전에 정의해야 한다. 이때 그 개수를 몇 개로 해야 할 것인지 문제가 될 수 있다. 이때 클러스터 개수를 여러 개 후보로 정하고 각 개수에 대해서 클러스터링 결과를 평가한다. 이때 평가에 활용된 지표(점수)를 최적화(최소화 또는 최대화)하는 클러스터 개수를 최종 선택한다.
3) 클러스터링(군집화) 결과를 비교

하나의 클러스터링(군집화) 알고리즘을 선택했다고 하자. 종종 여러 초기값의 대하여 클러스터링 결과를 비교하기 위하여 클러스터링(군집화) 평가가 필요하다.
이제 클러스터링(군집화)을 해야 할 필요성을 느꼈을 것이다.
그러면 이를 평가하기 위한 지표에는 무엇이 있을까?
Dunn Index와 Silhouette Index라는 것이 있다.
이에 대해 알아보자.
2. Dunn Index
Dunn Index는 클러스터링(군집화) 알고리즘(특히 K-Means 알고리즘) 결과를 평가하는 유명한 지표이며 클러스터 내(Intra-Cluster) 최대 거리에 대한 클러스터 간(Inter-Cluster) 최소 거리의 비로 나타낸다. 수식으로 표현하면 다음과 같다.
고정된 클러스터 개수 $K$에 대하여 Dunn Index $DI_K$는 다음과 같이 정의한다.
$$DI_K= \frac{ \underset{1\leq i \leq j \leq K}{\min} \delta (C_i, C_j)}{\underset{1\leq k\leq K}{\max}\Delta_k}$$
여기서 $\delta (C_i, C_j)$는 $i$번째 클러스터와 $j$번째 클러스터의 거리, $\Delta_k$는 $k$번째 클러스터 내 거리이다. 앞으로 특별한 언급이 없으면 $d$는 Euclidean Distance로 하겠다.
Dunn Index 같은 경우 그 값이 클수록 클러스터링(군집화)이 잘되었다고 판단한다.
이때 클러스터 간 거리 $\delta$와 클러스터 내 거리$\Delta$는 여러 가지로 정의할 수 있는데 이에 대해서 알아보자. 그런 다음 Dunn Index를 파이썬으로 구현해보겠다.
1) 클러스터 내(Intra-Cluster) 거리 측도
2) 클러스터 간(Inter-Cluster) 거리 측도
1) 클러스터 내(Intra-Cluster) 거리 측도
클러스터 내(Intra-Cluster) 거리는 한 군집 내의 데이터 간 거리를 나타낸 것으로 한 군집 내 변동성을 측정한 것이라고 할 수 있다. 클러스터 내(Intra-Cluster) 거리 측도는 여러 가지가 있으며 여기에서는 3가지를 살펴보겠다.
a. Complete Diameter Distance

Complete Diameter Distance는 다음과 같이 정의한다.
$$\Delta (C) = \max \left \{ d(a_i, a_j) \right \}$$
여기서 $d$는 거리 측도(예 : Euclidean Distance)이며 $a_i, a_j \in C$인 데이터 포인트, 즉 클러스터 $C$에 포함된 데이터 포인트들이다.
Complete Diameter Distance의 단점은 이상치(Outlier)의 영향을 많이 받을 수 있다. 또한 모든 조합의 거리를 다 살펴봐야 하므로 계산량이 많다.
이를 파이썬(Python)으로 구현해보자. 먼저 샘플 데이터를 만들어준다.
import numpy as np
import random
import matplotlib.pyplot as plt
from itertools import combinations
np.random.seed(100)
num_data = 20
x1 = np.linspace(0.3,0.7,num_data)
error = np.random.normal(1,0.5,num_data)
x2 = 1.5*x1+2+error
X = np.stack([x1, x2], axis=1)
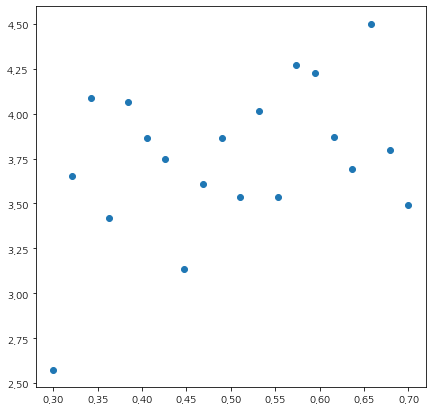
분포를 살펴보자.
fig = plt.figure(figsize=(7,7))
fig.set_facecolor('white')
plt.scatter(x1, x2, color='k')
plt.xlabel('x1')
plt.ylabel('x2')
plt.show()
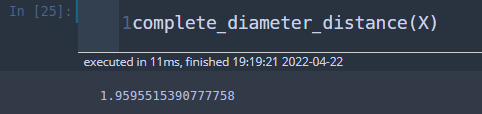
이제 Complete Diameter Distance를 계산하는 함수를 만들어보자. 먼저 itertools를 이용하여 데이터 매트릭스 행 인덱스 2개의 모든 조합을 뽑는다. 각 조합마다 거리를 계산하고 이를 리스트에 저장한 뒤 최대값을 리턴하면 된다.
def complete_diameter_distance(X):
res = []
for i, j in combinations(range(X.shape[0]),2):
a_i = X[i, :]
a_j = X[j, :]
res.append(np.linalg.norm(a_i-a_j))
return np.max(res)
b. Average Diameter Distance
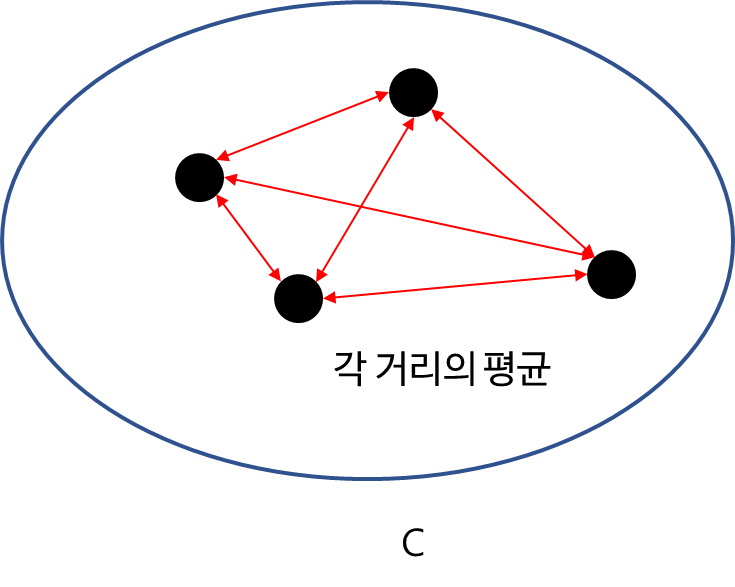
Complete Diameter Distance의 단점인 이상치 영향을 평균으로 어느 정도 상쇄시키고자 Average Diameter Distance를 생각할 수 있으며 그 정의는 다음과 같다.
$$\Delta (C) = \frac{1}{N_C\times (N_C - 1)}\sum_{i\neq j}d(a_i, a_j)$$
여기서 $N_C$는 $C$ 클러스터에 포함된 데이터 개수이다.
물론 Average Diameter Distance도 이상치에 영향을 받을 수 있다. 또한 조합의 수만큼의 계산량이 필요하므로 계산량이 상당하다.
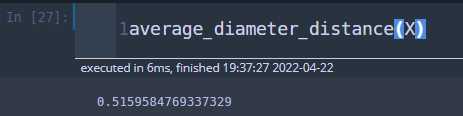
이제 파이썬(Python)으로 구현해보자. Average Diameter Distance 정의는 결국 모든 조합 거리의 평균이라는 점을 주목하자. 그러면 결국 모든 거리를 저장하는 리스트를 만든 뒤 평균을 계산하면 끝난다.
def average_diameter_distance(X):
res = []
for i, j in combinations(range(X.shape[0]),2):
a_i = X[i, :]
a_j = X[j, :]
res.append(np.linalg.norm(a_i-a_j))
return np.mean(res)
c. Centroid Diameter Distance
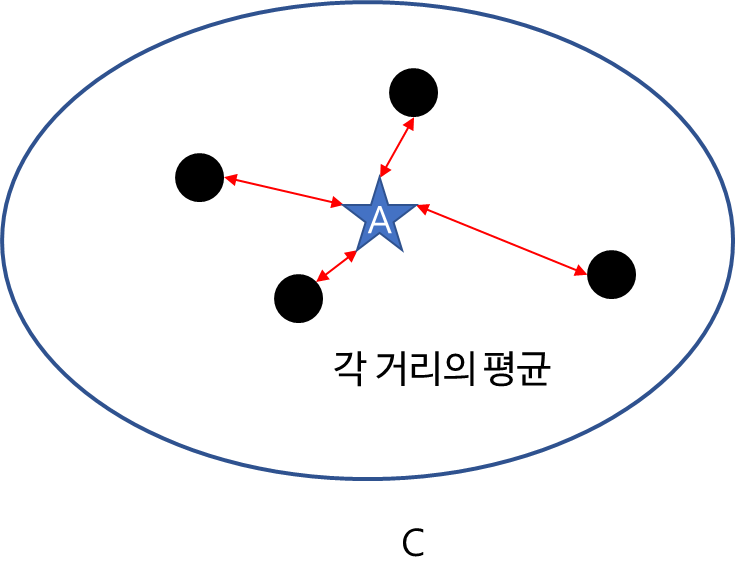
Complete Diameter Distance, Average Diameter Distance의 계산량을 완화시킨 Centroid Diameter Distance는 클러스터 중심과 데이터 간 거리들의 평균이다. 정의는 아래와 같다.
$$\Delta (C) = \frac{2}{N_C}\sum_{i=1}^{N_c}d(a_i, A)$$
여기서 $A$는 클러스터 $C$의 중심이다. 이때 중심은 특별한 말이 없으면 평균으로 하겠다.
Centroid Diameter Distance는 중심(평균) 계산과 각 거리 간의 평균을 계산하면 되므로 대략 $2N_C$ 만큼의 계산량이 필요하다. 앞서 살펴본 거리는 $N_C(N_C-1)/2$ 정도의 계산이 필요하다는 것을 감안하면 굉장히 적은 계산량이다.
하지만 Centroid Diameter Distance 또한 이상치에 영향을 받을 수 있다.
아래 코드는 파이썬으로 Centroid Diameter Distance를 계산하는 함수이다.
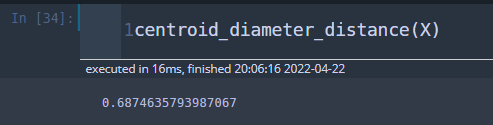
def centroid_diameter_distance(X):
center = np.mean(X, axis=0)
res = 2*np.mean([np.linalg.norm(x-center) for x in X])
return res
2) 클러스터 간(Inter-Cluster) 거리 측도
이번엔 클러스터 간(Intra-Cluster) 거리는 두 군집 간의 거리를 측정한 것으로 볼 수 있다. 거리 측도에는 무엇이 있는지 살펴보자.
a. Single Linkage Distance
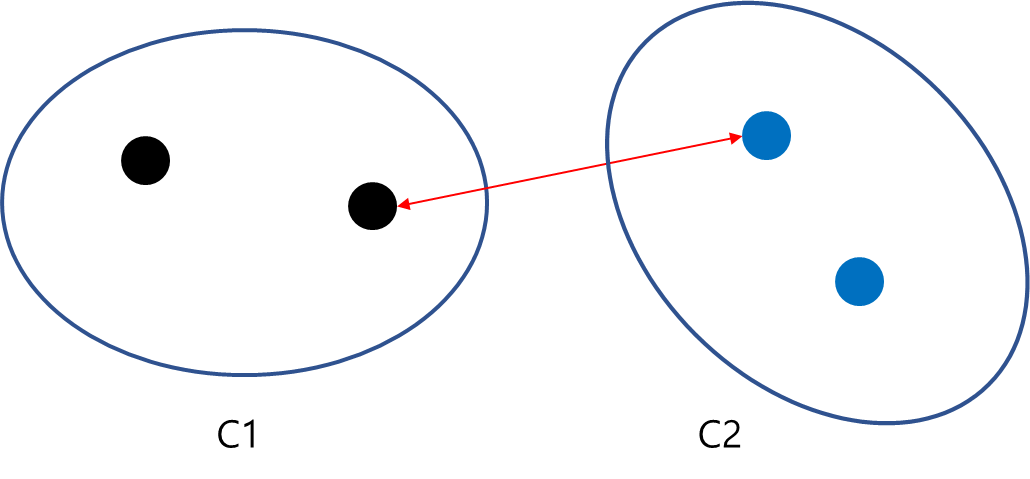
Single Linkage Distance는 서로 다른 두 클러스터의 속하는 데이터들의 거리 중에서 최소값을 의미하며 다음과 같이 정의한다.
$$\delta (C_1, C_2) = \min d(a , b), \text{ where } a \in C_1, b\in C_2$$
파이썬으로 구현해보자.
먼저 샘플 데이터를 만들어 준다.
np.random.seed(100)
x11 = np.linspace(0.3,0.7,20)
label1 = [0]*len(x11)
x12 = np.linspace(1.3,1.8,15)
label2 = [1]*len(x12)
error = np.random.normal(1,0.5,35)
x1 = np.concatenate((x11,x12),axis=None)
x2 = 1.5*x1+2+error
labels = label1+label2
X = np.stack((x1, x2), axis=1)
labels = np.array(labels)
X1 = X[np.where(labels==0)[0], :]
X2 = X[np.where(labels==1)[0], :]
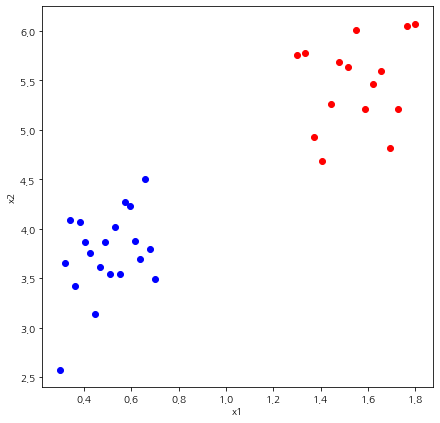
그리고 어떻게 생겼는지 확인하기 위해 시각화해본다.
fig = plt.figure(figsize=(7,7))
fig.set_facecolor('white')
for i, x in enumerate(X):
if labels[i] == 0:
plt.scatter(x[0], x[1], color='blue')
else:
plt.scatter(x[0], x[1], color='red')
plt.xlabel('x1')
plt.ylabel('x2')
plt.show()
이제 Single Linkage Distance를 계산하는 함수를 구해보자. 무식하게 두 개의 루프를 이용하여 두 군집 간 모든 데이터 거리를 계산하고 최소값을 리턴한다.
def single_linkage_distance(X1, X2):
res = []
for x1 in X1:
for x2 in X2:
res.append(np.linalg.norm(x1-x2))
return np.min(res)

b. Complete Linkage Distance
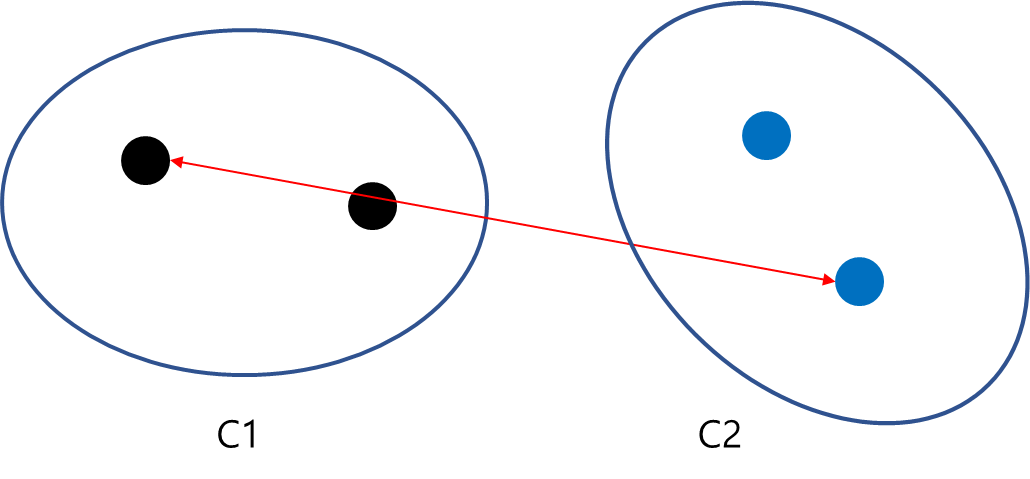
Complete Linkage Distance는 반대의 개념으로 서로 다른 두 클러스터의 속하는 데이터들의 거리 중에서 최대값을 의미하며 다음과 같이 정의한다.
$$\delta (C_1, C_2) = \max d(a , b), \text{ where } a \in C_1, b\in C_2$$
파이썬을 이용하여 Complete Linkage Distance를 계산하는 함수를 만들어보자.
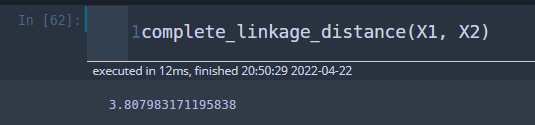
def complete_linkage_distance(X1, X2):
res = []
for x1 in X1:
for x2 in X2:
res.append(np.linalg.norm(x1-x2))
return np.max(res)
c. Average Linkage Distance
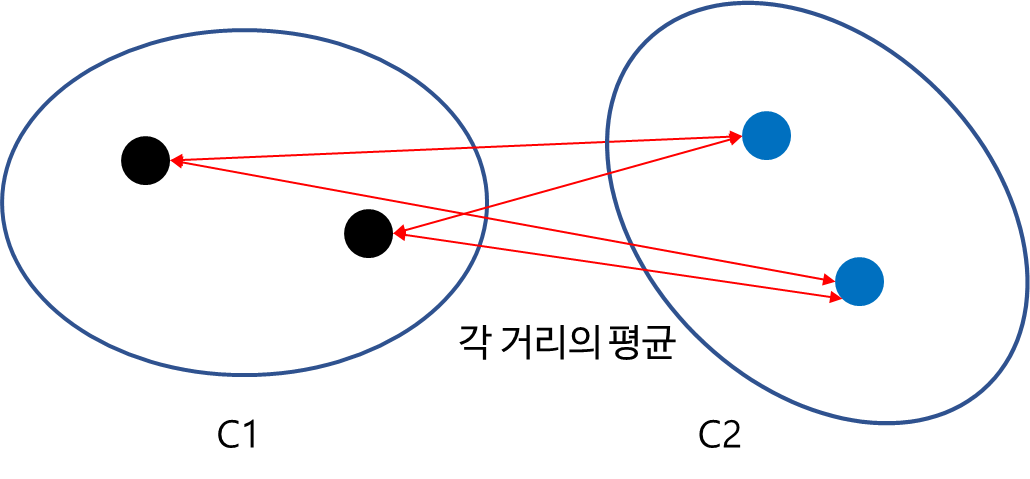
Average Linkage Distance는 두 군집 간 모든 데이터의 거리를 계산한 뒤 평균을 계산한 거리이다.
$$\delta (C_1, C_2) = \frac{1}{N_{C_1}N_{C_2}}\sum_{i=1}^{N_{C_1}}\sum_{j=1}^{N_{C_2}}d(a_i, b_j)$$
여기서 $a_i \in C_1, b_j \in C_2$ 이고 $N_{C_1}, N_{C_2}$는 각각 클러스터 $C_1, C_2$에 포함된 데이터 개수이다.
파이썬을 이용하여 Average Linkage Distance를 계산하는 함수를 만들어보자.
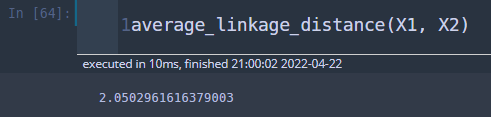
def average_linkage_distance(X1, X2):
res = []
for x1 in X1:
for x2 in X2:
res.append(np.linalg.norm(x1-x2))
return np.mean(res)
d. Centroid Linkage Distance
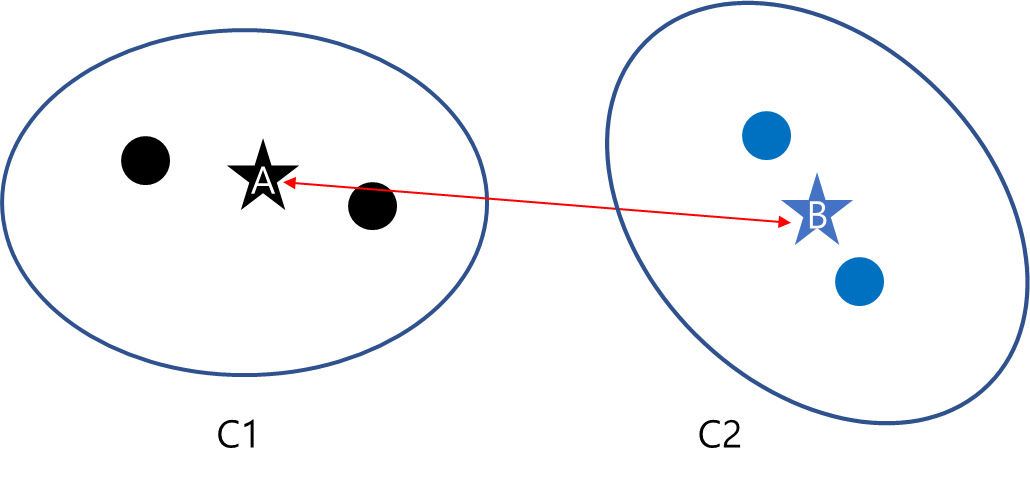
Centroid Linkage Distance은 각 군집의 중심 간 거리를 계산한다.
$$\delta (C_1, C_2)= d(A, B)$$
여기서 $A, B$는 각각 클러스터 $C_1, C_2$의 중심값(보통 평균)이다.
Python(파이썬)을 이용하여 Centroid Linkage Distance를 계산하는 함수를 만들어보자.
def centroid_linkage_distance(X1, X2):
center1 = np.mean(X1, axis=0)
center2 = np.mean(X2, axis=0)
return np.linalg.norm(center1-center2)
e. Average of Centroids Linkage Distance
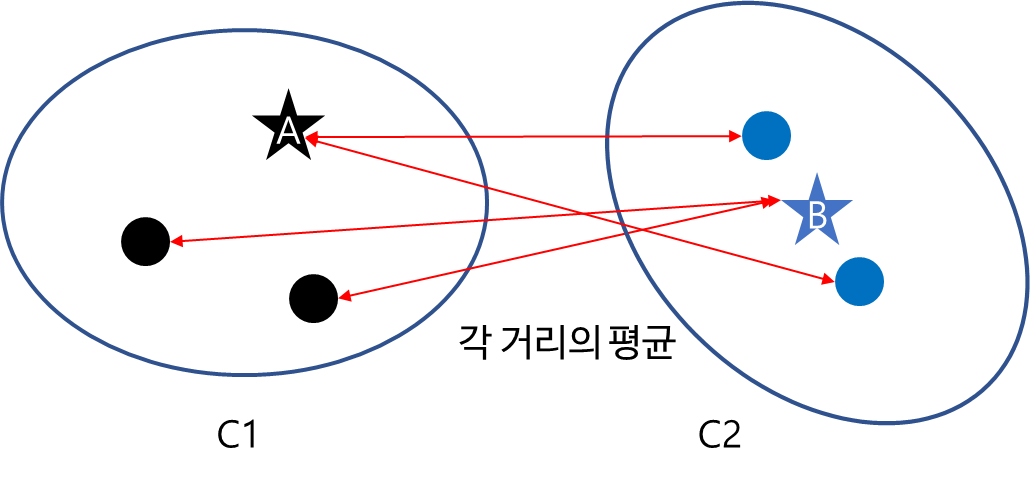
Average of Centroids Linkage Distance은 한 클러스터(군집)의 중심과 다른 클러스터(군집)의 데이터 포인트 간 거리를 계산하여 평균을 구하게 된다.
$$\delta (C_1, C_2) = \frac{1}{N_{C_1}+N_{C_2}}\left\{ \sum_{i=1}^{N_{C_1}}d(a_i, B)+\sum_{j=1}^{N_{C_2}}d(b_j, A) \right\}$$
Python(파이썬)을 이용하여 Average of Centroids Linkage Distance를 계산하는 함수를 만들어보자.
def average_of_centroids_linkage_distance(X1, X2):
center1 = np.mean(X1, axis=0)
center2 = np.mean(X2, axis=0)
res = []
for x1 in X1:
res.append(np.linalg.norm(x1-center2))
for x2 in X2:
res.append(np.linalg.norm(x2-center1))
return np.mean(res)
3) Dunn Index 파이썬 구현
이제 클러스터 내(Intra-Cluster) 거리 측도, 클러스터 간(Inter-Cluster) 거리 측도의 종류를 알아보았으니 이를 이용하여 Dunn Index를 파이썬으로 구현해보자.
샘플 데이터를 먼저 만들어 준다.
np.random.seed(100)
num_data = 50
x11 = np.linspace(0.3,0.7,20)
label1 = [0]*len(x11)
x12 = np.linspace(1.3,1.8,15)
label2 = [1]*len(x12)
x13 = np.linspace(2.4,3,15)
label3 = [2]*len(x13)
x1 = np.concatenate((x11,x12,x13),axis=None)
error = np.random.normal(1,0.5,num_data)
x2 = 1.5*x1+2+error
X = np.stack((x1, x2), axis=1)
labels = np.array(label1+label2+label3)
fig = plt.figure(figsize=(7,7))
fig.set_facecolor('white')
for i, x in enumerate(X):
if labels[i] == 0:
plt.scatter(x[0], x[1], color='blue')
elif labels[i] == 1:
plt.scatter(x[0], x[1], color='red')
else:
plt.scatter(x[0], x[1], color='green')
plt.xlabel('x1')
plt.ylabel('x2')
plt.show()
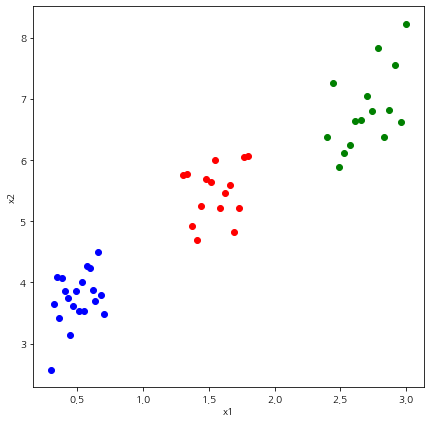
위와 같은 클러스터링 결과에 대하여 Dunn Index를 계산하는 함수를 만들어보자. 먼저 클러스터 내(Intra-Cluster)와 클러스터 간(Inter-Cluster) 거리 측도를 계산할 함수를 선정하고 $DI_K$를 계산한다. 이때 클러스터 내 거리 계산 시 데이터가 2포인트 이상있어야 하며 그렇지 않은 경우는 0으로 세팅했다.
def get_Dunn_index(X, labels, intra_cluster_distance_type,
inter_cluster_distance_type):
intra_cdt_dict = {
'cmpl_dd':complete_diameter_distance,
'avdd' : average_diameter_distance,
'cent_dd' : centroid_diameter_distance
}
inter_cdt_dict = {
'sld' : single_linkage_distance,
'cmpl_ld' :complete_linkage_distance,
'avld': average_linkage_distance,
'cent_ld' : centroid_linkage_distance,
'av_cent_ld' : average_of_centroids_linkage_distance
}
# intra cluster distance
intra_cluster_distance = intra_cdt_dict[intra_cluster_distance_type]
# inter cluster distance
inter_cluster_distance = inter_cdt_dict[inter_cluster_distance_type]
# get minimum value of inter_cluster_distance
res1 = []
for i, j in combinations(np.unique(labels),2):
X1 = X[np.where(labels==i)[0], :]
X2 = X[np.where(labels==j)[0], :]
res1.append(inter_cluster_distance(X1, X2))
min_inter_cd = np.min(res1)
# get maximum value of intra_cluser_distance
res2 = []
for label in np.unique(labels):
X_target = X[np.where(labels==label)[0], :]
if X_target.shape[0] >= 2:
res2.append(intra_cluster_distance(X_target))
else:
res2.append(0)
max_intra_cd = np.max(res2)
Dunn_idx = min_inter_cd/max_intra_cd
return Dunn_idx
이제 클러스터링(군집화) 결과에 대하여 평가를 해보자. Dunn Index를 계산하면 된다.
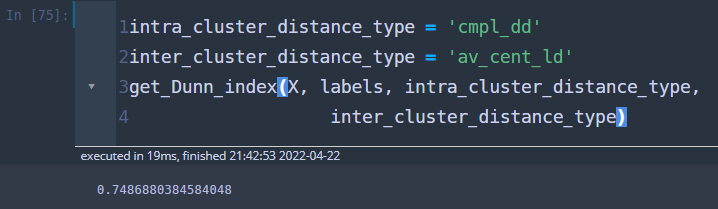
0.7486 정도가 나왔다. 1에 가까운 값일수록 클러스터링이 잘되었다고 하는데 이 정도면 나쁘지 않은 것 같다.
이번 포스팅에서는 클러스터링(군집화) 결과를 평가하는 지표로 Dunn Index를 알아보았다. 다음 포스팅에서는 다른 평가 지표로 Silhouette Index에 대해서 알아보겠다.
참고자료
Dunn Index for K-Means Clustering Evaluation
- https://python-bloggers.com/2022/03/dunn-index-for-k-means-clustering-evaluation/

'통계 > 머신러닝' 카테고리의 다른 글
| 13. Box-Cox Transformation(변환)에 대해서 알아보자 with Python (385) | 2022.05.01 |
|---|---|
| 12. Gaussian Mixture Model(가우시안 혼합 모형) 클러스터링(군집 분석)에 대해서 알아보자 with Python (408) | 2022.04.25 |
| 11. K-Means 클러스터링(Clustering, 군집화)에 대해서 알아보자 with Python (410) | 2022.04.21 |
| 10. 가지치기(Pruning)에 대해서 알아보자 with Python (1267) | 2021.07.05 |
| 9. 의사결정나무(Decision Tree) 에 대해서 알아보자 with Python (1275) | 2021.06.10 |





댓글